在植物基因组这片广袤的“荒原”中,转座子(transposon)宛如不安分的“游牧者”,随时可能跳跃、插入,扰乱基因组的秩序。在玉米、稻米等作物中,转座子序列占据了基因组的60%至80%。尽管大多数转座子在正常情况下被沉默,但在环境胁迫或表观调控失衡时,它们可能被重新激活,干扰基因表达甚至威胁基因组稳定。为维持基因组的稳定与沉默,植物演化出多层次表观遗传调控机制,依靠H3K9me2、H3K27me1等组蛋白修饰建立异染色质状态,限制转座子转录。然而,RNA层面的表观修饰是否也参与调控染色质结构与转录活性,长期以来仍是未解之谜。
2025年10月,Nature Plants在线发表了北京大学化学与分子工程学院贾桂芳教授团队的最新研究成果,论文题为“RNA m⁶A regulates the transcription and heterochromatin state of retrotransposons in Arabidopsis”。该研究首次揭示了m⁶A修饰通过跨越RNA与染色质层面的“反向调控”机制,调节植物逆转录转座子的转录活性与异染色质状态。
研究团队构建了适用于植物的高分辨率染色质相关RNA m⁶A测序技术(caRNA m⁶A-seq),系统绘制了拟南芥染色质RNA的m⁶A图谱。结果显示,m⁶A修饰在逆转录转座子RNA上显著富集,主要由甲基转移酶复合物MTA/FIP37/VIR“写入”,并由核内m⁶A结合蛋白CPSF30-L识别。当这些转座子RNA的m⁶A水平下降时,其转录显著增强。结合核内RNA半衰期测定与新生RNA标记实验,研究表明m⁶A主要通过抑制转录速率而非影响RNA稳定性实现转录沉默。
进一步研究发现,m⁶A富集区域常伴随高水平的异染色质修饰H3K9me2和H3K27me1,而在m⁶A甲基转移酶或识别蛋白突变体中,这两种修饰显著下降,对应区域的转座子及邻近基因被激活。机制分析揭示,CPSF30-L不仅直接结合m⁶A标记RNA,还与组蛋白甲基转移酶SUVH4/5/6和ATXR5/6相互作用,促进H3K9me2和H3K27me1沉积,从而维持局部异染色质结构与基因沉默。与此同时,研究鉴定到另一核内m⁶A结合蛋白ECT12,CPSF30-L与ECT12可形成复合物协同识别m⁶A,共同维持逆转录转座子的转录沉默与异染色质状态(图1)。
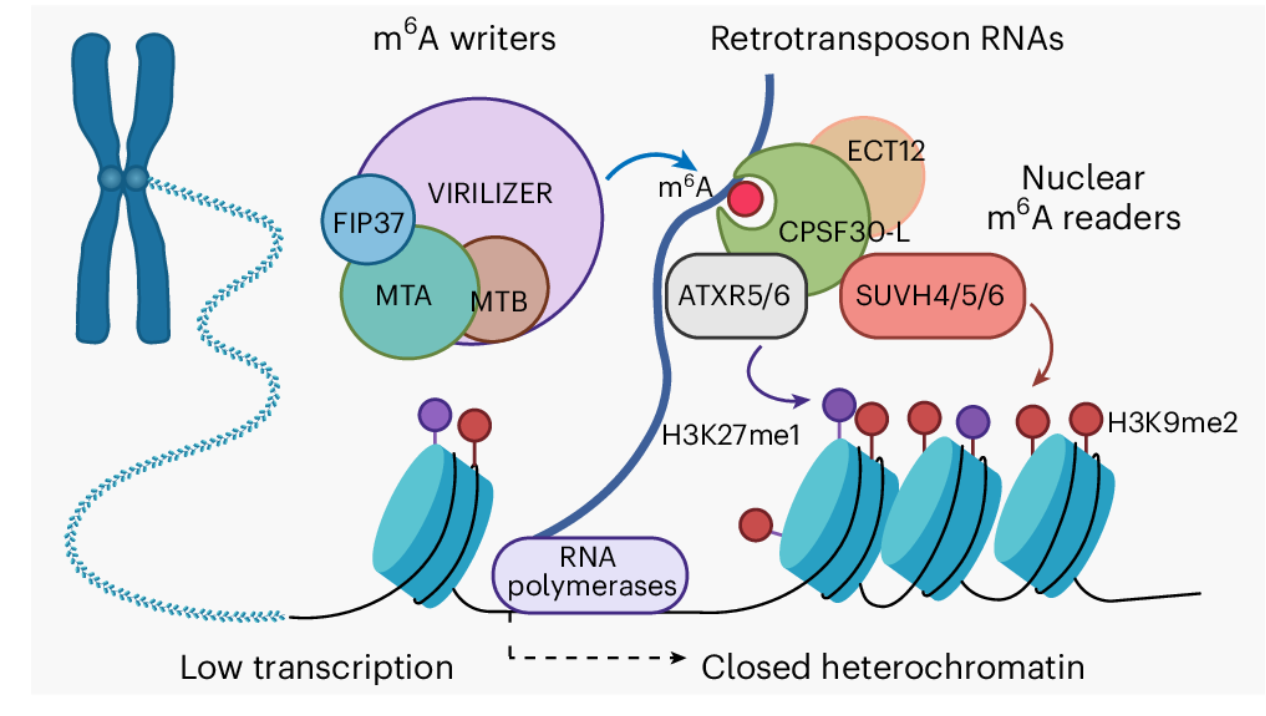
图1|m⁶A 调控逆转录转座子转录及异染色质状态的分子机制模型图。
本研究首次揭示了从RNA修饰到染色质状态的跨层级表观遗传调控通路:m⁶A甲基化—识别蛋白复合物—组蛋白修饰—染色质状态—转录沉默。这种从RNA到染色质的反向调控模式,拓展了对表观转录组(epitranscriptome)与表观基因组(epigenome)互作关系的理解,也为通过RNA修饰精准调控作物基因表达、增强抗逆性和改良性状提供了新思路。
北京大学化学与分子工程学院、北大-清华生命科学联合中心贾桂芳教授为论文通讯作者。贾桂芳课题组副研究员宋培哲、2021级博士研究生才智赫、2020级毕业博士研究生苏比丁·塔依尔为论文共同第一作者。贾桂芳课题组田恩麟、陈子昕博士、余珂淼及刘骊翔等参与了研究工作。该研究得到农业部科技创新2030项目、国家重点研发计划、国家自然科学基金、北大-清华生命联合研究中心、核糖核酸北京研究中心的资助。
排版:高杨
审核:牛林,刘志博