蛋白质复合物结构预测对于理解生物活动和推进药物开发至关重要。实验方法可以为蛋白质复合物提供结构洞见,但获得的知识通常是稀疏或近似的。需要一个通用工具来整合有限的实验信息以实现高通量和准确的预测。在这里,我们介绍了GRASP,它可以高效灵活地整合多种形式的实验信息。GRASP在处理模拟和真实世界的实验限制方面优于现有工具,包括交联、共价标记、化学位移扰动和深度突变扫描的限制。例如,GRASP在预测抗原-抗体复合物结构方面表现出色,甚至在使用实验深度突变扫描或共价标记限制时超越了AlphaFold3。GRASP在受限结构预测中的准确性和灵活性之外,其整合多种形式限制的能力还支持整合建模。该研究还使用了先前报道的大规模线粒体原位交联数据进行高通量互作组建模,展示了其在模拟细胞条件下的蛋白质结构互作网络方面的潜力。
研究背景
1.研究问题:这篇文章要解决的问题是如何有效地整合多种实验信息以辅助或增强蛋白质复合物结构预测。蛋白质复合物结构对于理解生物活动和推动药物开发至关重要,但现有的实验方法提供的结构信息往往是稀疏或有噪音的,如何充分利用这些多样的信息提高复合物结构建模的精度成为计算与实验科学家共同关注的问题。
2.研究难点:该问题的研究难点包括:处理不同来源和形式的实验数据(如化学交联、共价标记、化学位移扰动和深度突变扫描)、提高通量、准确性和对噪音的鲁棒性、以及整合多种约束信息以实现整合建模。
3.相关工作:现有的工具如AlphaFold系列、ESMFold等在蛋白质复合物结构预测中的准确性有限。其他工具如AlphaLink、ColabDock、HADDOCK和ClusPro虽然支持实验约束,但在处理特定类型的约束或长序列时存在局限性。
研究方法
这篇论文提出了GRASP模型,用于解决蛋白质复合物结构预测问题。具体来说,
模型架构:GRASP将残基对约束(RPR)和界面约束(IR)直接整合到AFM中进行蛋白质复合物结构预测。RPR来自实验方法如交联质谱和核磁谱,提供了残基对之间的距离约束;IR来自实验方法如共价标记、化学位移扰动和深度突变扫描,指示残基是否位于界面。
约束整合:RPR通过Evoformer模块的MSA偏执项和结构模块的不变点注意力(IPA)偏执项整合到GRASP中;IR则通过相对位置编码和Evoformer及不变点注意力模块中的界面融合块引入。
损失函数与训练:在原始损失函数中添加了四个与约束相关的损失项,并分别从AFM的v.2.3 Model-1检查点(checkpoint)以及自行从头训练的MEGAFold-Multimer检查点出发,使用64块Ascend-910A分别进行了22,000步微调训练,获得了共5组模型参数。
迭代噪声过滤:在推理过程中应用迭代噪声过滤策略,以减少错误约束的影响。
实验设计
1.数据集准备:训练数据集基于团队之前整理公开的PSP数据集,包含570,000个真实结构集和760,000个蒸馏集。自制的测试数据集从发布时间严格晚于训练集的PDB结构数据进行构建,并经过复合物重复度、质量、难度的多重筛选,最终包含313个复合物的713个界面。
2.模拟约束采样:对313个蛋白质复合物进行随机分组,每组采样不同数量的RPR和IR。模拟的XL数据集在测试数据集上分别进行约束覆盖率1%、2%和5%的XL模拟采样并去除约束错误率过高的样本,包含294个复合体。模拟的抗原-抗体DMS数据集基于两个抗原-抗体数据集构建,每侧随机采5-10个IR模拟DMS数据提供的界面信号。
3.实验数据集:构建了包含单一实验约束类型的单类型实验数据集,包括九个XL、五个CL和四个CSP案例。实验DMS数据集包含84个SARS-CoV-2 RBD靶向单克隆抗体的深度突变扫描谱。线粒体数据集包含144个蛋白质相互作用对。
4.整合建模数据集:包括四个复合物案例,每个案例均使用多种实验信息进行整合建模。使用的实验约束类型分别为:突变+化学交联+低分辨率冷冻电镜、二硫键交联+镉(II)-半胱氨酸桥接+双突变循环分析、化学交联+PRE、化学交联+冷冻电镜。
自制测试数据集上的性能:在没有约束的情况下,AF3的表现略优于AFM;但随着约束数量的增加,基于约束的方法的预测准确性普遍提高。GRASP在所有方法中始终排名第一,在不同数量的IR和接触RPR下表现出最大的增益,并且构建了表面可及性、结合能和界面氢键数和真实结构近似、有物理意义的界面。
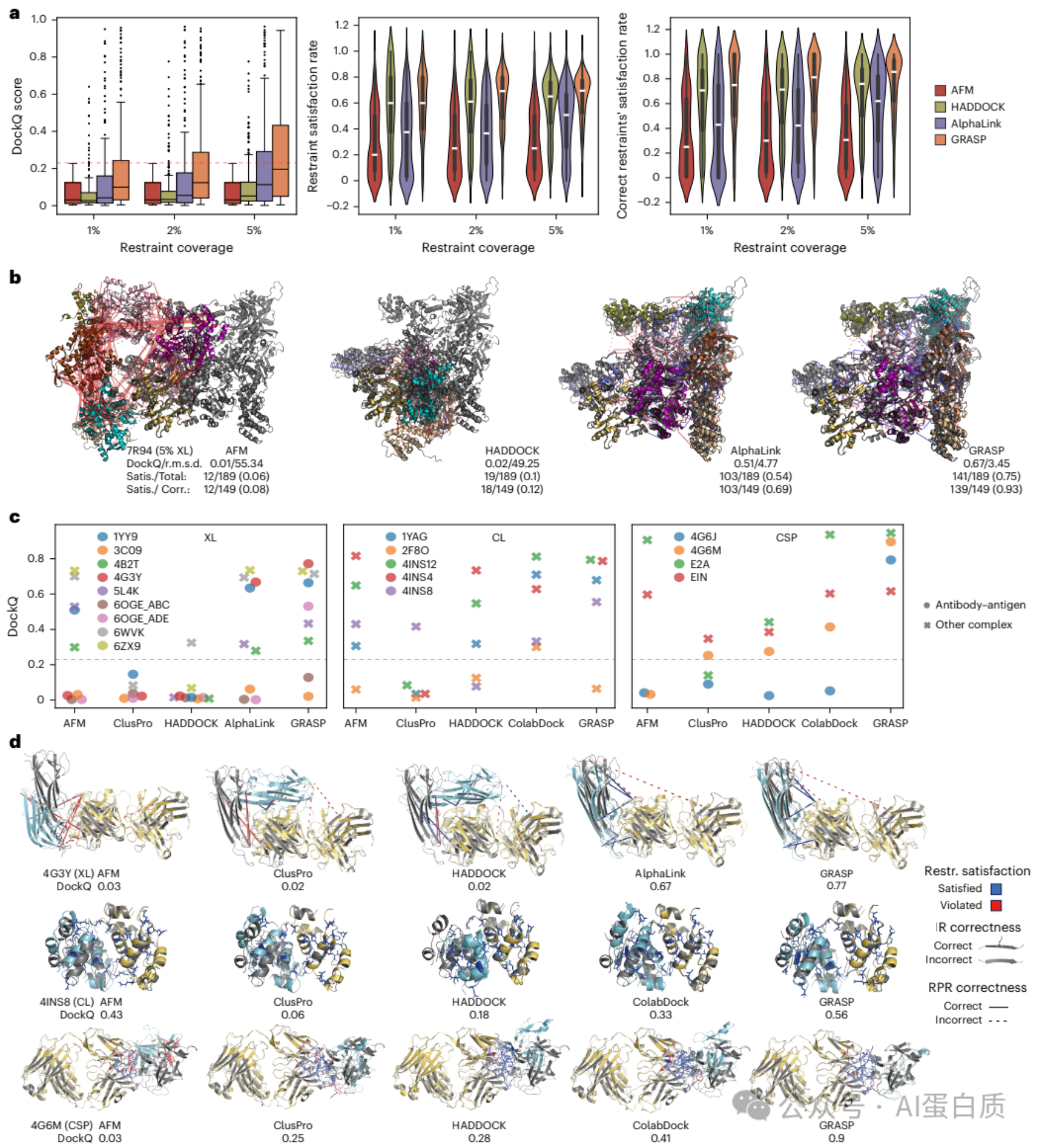
XL、CL 和 CSP 约束下的性能:GRASP在所有XL约束覆盖率下的表现均优于 HADDOCK 和 AlphaLink。例如,在1%的约束覆盖率下,GRASP 的平均 DockQ 为0.18,而 HADDOCK 和 AlphaLink 分别为0.06和0.12。在 CL 和 CSP 真实实验约束下 GRASP 的表现依然稳定优于其他方法。
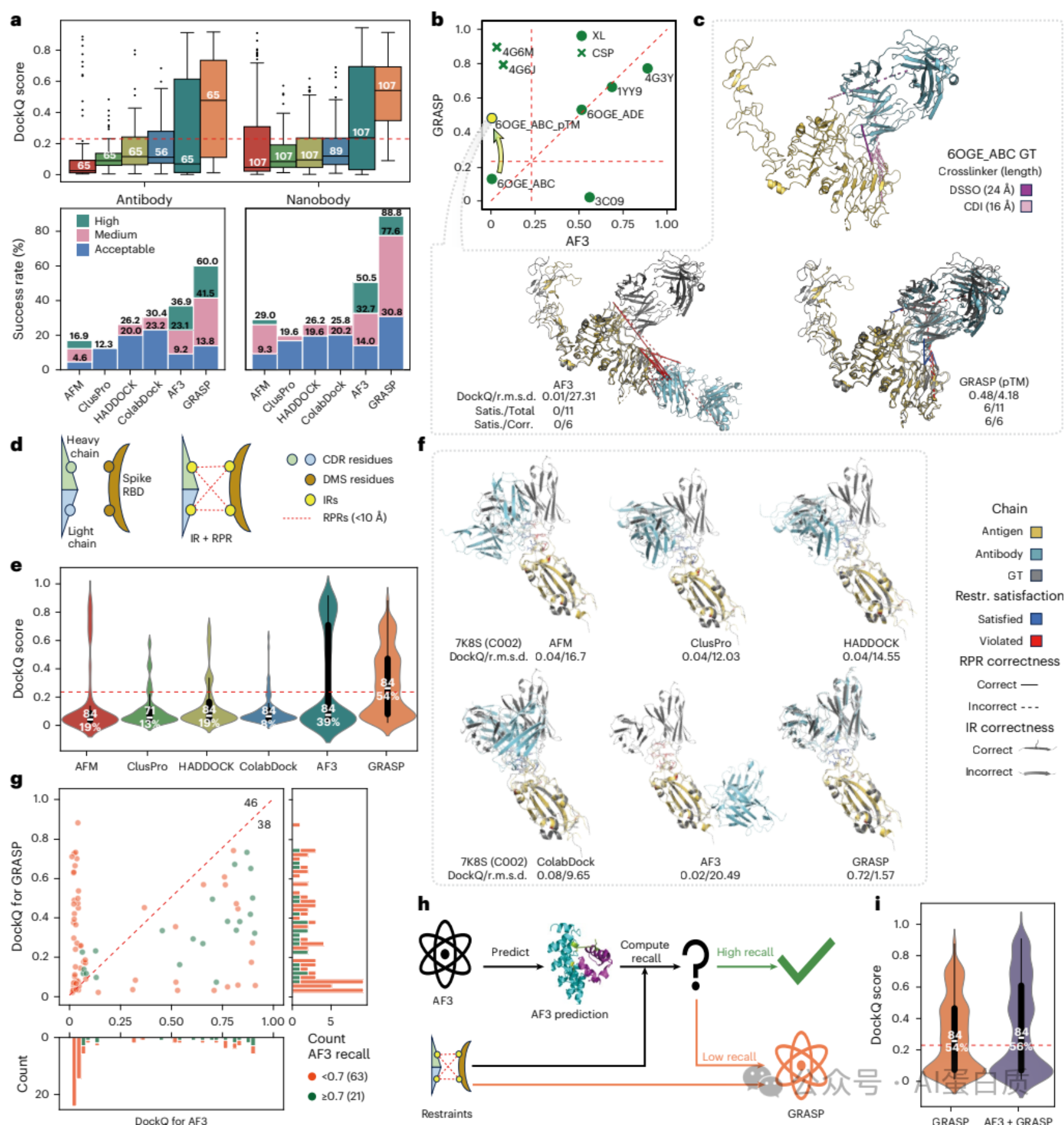
抗体-抗原复合物预测:GRASP 在 BM5.5 数据集和严格防止数据泄露的Hitawala-Gray数据集上的表现优于所有其他方法,在后者上中位 DockQ 得分为 0.477(常规抗体)和 0.541(纳米抗体),成功率分别为60.0%和88.8%。在CSP 和XL 实验数据集上,GRASP 的成功率为5/7,优于AF3的4/7。在84个样本的DMS实验数据集上,GRASP 的表现超越了所有其它方法,成功率为53.8%,AF3则为39.3%。作者进一步提出GRASP和AF3结合的联合策略,可以进一步提高成功率。
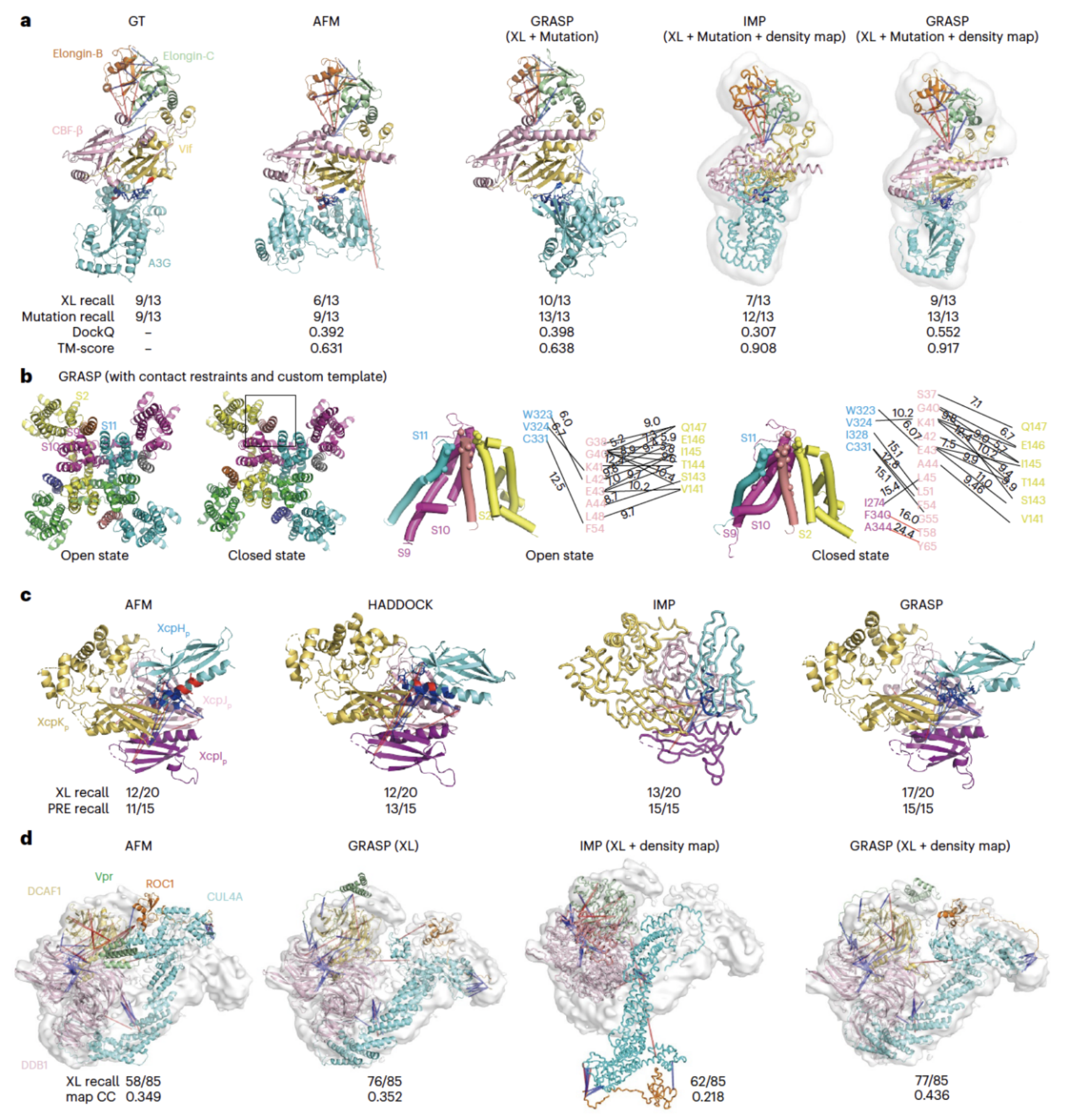
整合建模:为利用更加丰富的实验数据进行整合建模,该研究提出了Combfit工具以间接整合冷冻电镜信息。GRASP通过结合冷冻电镜密度图、XL、NMR PRE、突变和半胱氨酸桥接等实验,展示了其在四个复杂约束建模任务中的潜力。例如,在 A3G-VCBC 系统中,GRASP 结合XL、突变数据和中低分辨率冷冻电镜数据后,满足所有界面约束和 9/13 交联约束,DockQ 为0.552。
该研究提出的GRASP模型通过直接整合多种实验约束信息,显著提高了蛋白质复合物结构预测的准确性和可靠性。GRASP在处理模拟和真实世界实验约束方面表现出色,特别是在抗体-抗原复合物预测中超越了现有的AlphaFold3。GRASP还可以作为整合建模工具,结合多种实验信息进行复杂的约束建模。未来的工作将进一步优化GRASP的集成策略,以解决更具挑战性的应用场景。
昌平实验室-北京大学联合培养博士生谢豫豪和北京大学博士生张成伟为本文的共同第一作者,北京大学、昌平实验室的高毅勤教授和昌平实验室刘思睿研究员为本文的通讯作者。作者们向K. Stahl教授、F. N. Hitawala 博士、杨开广研究员、朱祯博士表示感谢。该工作得到了科技部、基金委、昌平实验室、北京大学前沿交叉学科研究院、北京大学生物医学前沿创新中心(BIOPIC)、北京大学生命科学学院、北京大学化学与分子工程学院,北京分子科学国家研究中心,新基石科学实验室、华为技术有限公司的支持。
原文链接:
nature.com/articles/s41592-025-02820-1
排版:高杨
审批:牛林,刘志博