生命如同一台精密的生物计算机,其核心编程语言由DNA分子中 A、T、C、G 四种碱基构成的64组密码子组成。这些遗传密码通过 RNA 介导的翻译过程,指导细胞利用20种标准氨基酸合成功能各异的蛋白质。突破这一天然遗传密码的限制,将开启生命编程的新维度,为生物医学领域带来革命性变革。
传统的 DNA 密码子扩展技术在真核生物系统中存在引发终止密码子通读等安全性问题1。这促使科学家将目光转向更具可塑性的 RNA 分子。RNA 不仅摆脱了 DNA 的复制与修复限制,其天然存在的 170 余种化学修饰更为遗传密码的工程化改造提供了丰富素材。
2025年6月25日,北京大学化学与分子工程学院的陈鹏团队和生命科学学院伊成器团队合作在 Nature 发表题为RNA codon expansion via programmable pseudouridine editing and decoding 的论文,通过 RNA 假尿嘧啶 Ψ 修饰的定点编程,首次创造并编码了三个 ΨCodon “密码子”,进一步筛选并获得了选择性解码 ΨCodon 的 tRNA 。该方法成功构建了一套基于RNA修饰的新型编程语言,突破了64种密码子和20种天然氨基酸的“中心法则”限制,可驱动活细胞将非天然氨基酸定点、精准融入蛋白质的生物合成,实现蛋白质在细胞内的化学“定制合成”,从而可实现对纷繁复杂的蛋白质“变体”的精准体内合成和原位研究。这不仅拓展了生命系统的设计空间,更为合成生物学和精准医学开辟了新路径。通过重构生命的信息处理原件,生命编程的基本范式正在被改写。

RNA 密码子拓展技术:编码和解码全新的RNA密码子
该研究通过开发新型 RNA 密码子系统,突破了传统 DNA 遗传密码的 64 种密码子限制,实现了对非天然氨基酸的精准编码与解码。研究团队采用序列特异性的假尿苷(Ψ)修饰技术,利用向导 RNA 在目标转录本的 UGA 终止密码子上引入 Ψ 修饰,成功构建了新型人工密码子"ΨGA"(这类修饰的终止密码子统称为 ΨCodon ),为蛋白质的定制合成和工程改造提供了全新的编码工具(图1)。
为实现 ΨCodon 的高效解码,研究者基于合成酶-tRNA 的晶体结构数据,构建了饱和单点突变文库,并通过筛选获得了具有高度密码子偏好性的 tRNA 变体(ΨGA-tRNAPyl)。该 tRNA 变体在多种报告系统中均表现出对 ΨGA 的特异性识别能力,确保其解码过程不会干扰天然 UGA 终止密码子的正常功能。最终,通过整合 ΨGA 编码模块与 ΨGA-tRNAPyl 解码模块,研究团队成功构建了 RCE(ΨGA) 系统,为人工设计功能性蛋白质开辟了新途径。
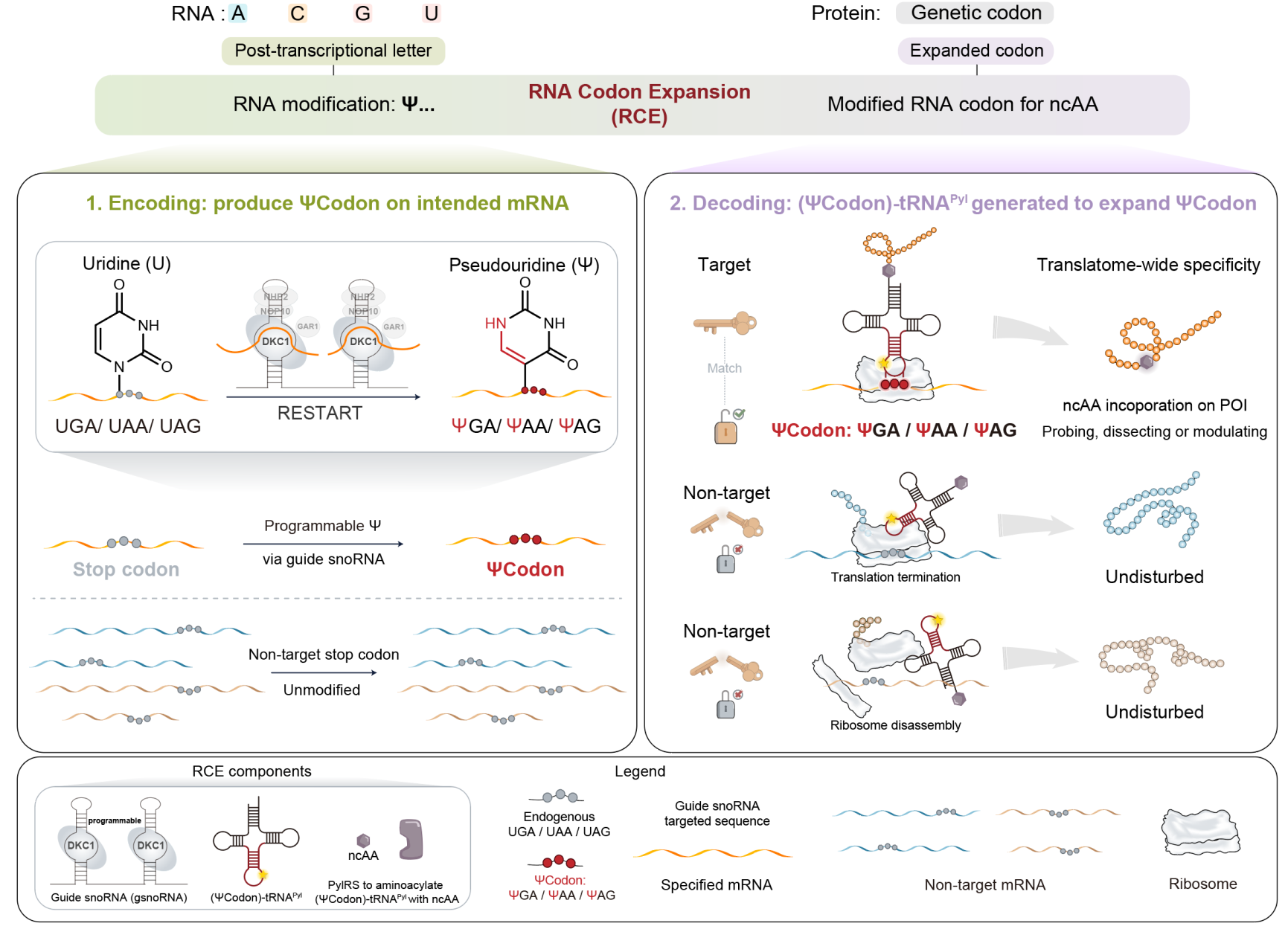
图1. RNA 密码子拓展策略示意图。该策略包含编码和解码两个核心过程,从而实现非天然氨基酸的特异性插入。
RCE系统具有更高的全局特异性
成功构建 RCE(ΨGA) 系统后,研究团队采用多种组学方式对该技术的全局特异性进行了系统性验证。首先,通过前期开发的Ψ修饰测序技术 "PRAISE",在全转录组水平检测发现该系统仅产生少量脱靶修饰,且未影响正常终止密码子。进一步,研究团队采用核糖体分析技术评估翻译组特异性,结果显示 RCE 系统能精准识别 ΨGA 密码子进行翻译,同时全转录组中 UGA 密码子的非特异性通读率显著低于传统遗传密码扩展技术(Genetic Code Expansion)。
为全面评估翻译产物的特异性,研究团队利用利用非天然氨基酸TetBu分子的四嗪基团,与反式环辛烯偶联的生物素分子的成键反应,实现了全蛋白质组范围内非天然氨基酸插入位点的精准检测。蛋白质组学分析表明,RCE系统的脱靶蛋白数量较传统GCE技术显著减少,GO功能富集分析进一步证实其对关键生物学通路无明显干扰(图2)。
综合转录组、翻译组和蛋白质组三个层面的分析结果,充分证明 RCE 技术通过 Ψ 修饰密码子的特异性编解码,实现了非天然氨基酸的高精准插入并显著降低了脱靶效应。
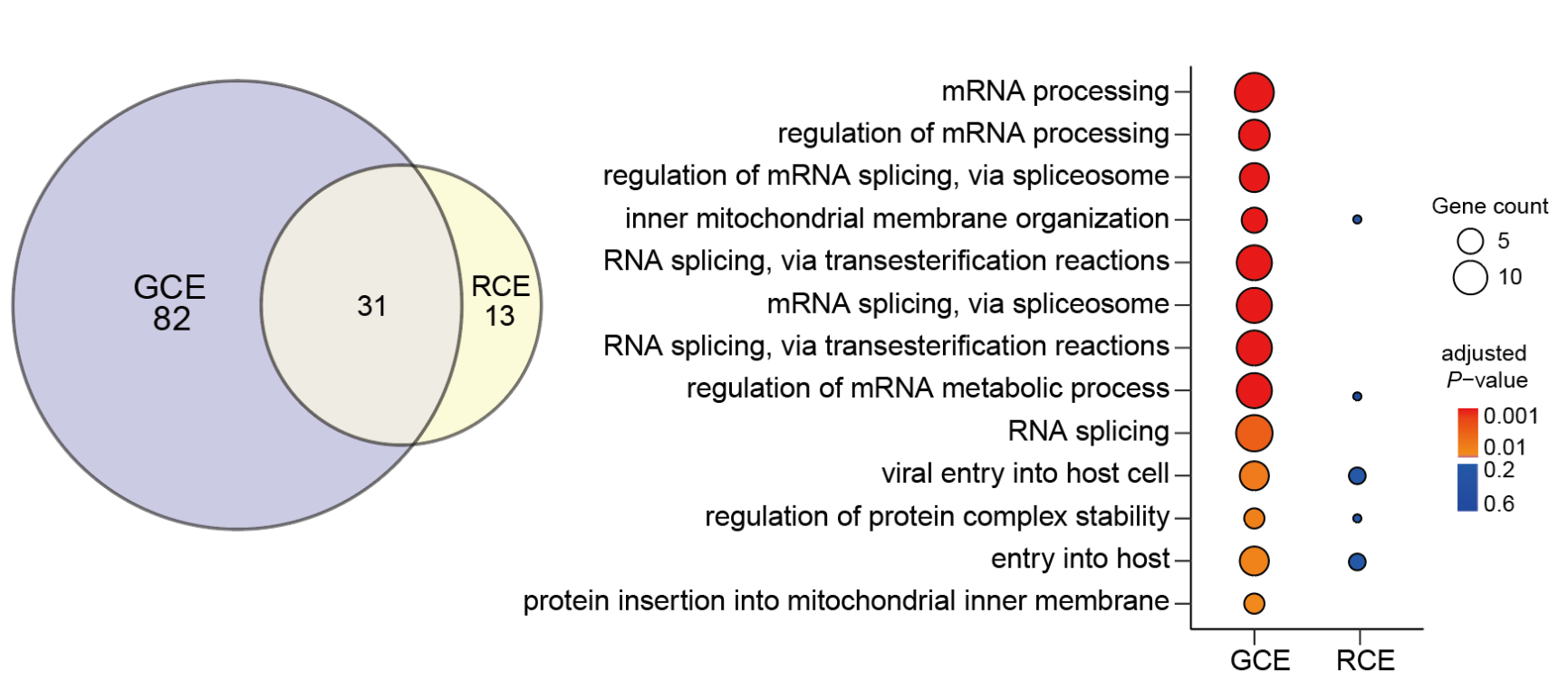
图2. 蛋白质组分析显示RCE系统在全蛋白质组中具有全局特异性。
RCE密码子的拓展
研究团队进一步拓展了 RCE 系统的密码子范围。通过采用与 ΨGA-tRNAPyl 类似的筛选策略,成功获得了针对 ΨAA 和 ΨAG 密码子的特异性 tRNA 解码器。实验验证表明,这三种 tRNA 解码器(ΨGA-tRNAPyl、ΨAA-tRNAPyl和ΨAG-tRNAPyl)表现出优异的正交性:每个解码器仅识别其对应的 ΨCodon ,而不会通读其他类型的 Ψ 修饰密码子(图3)。
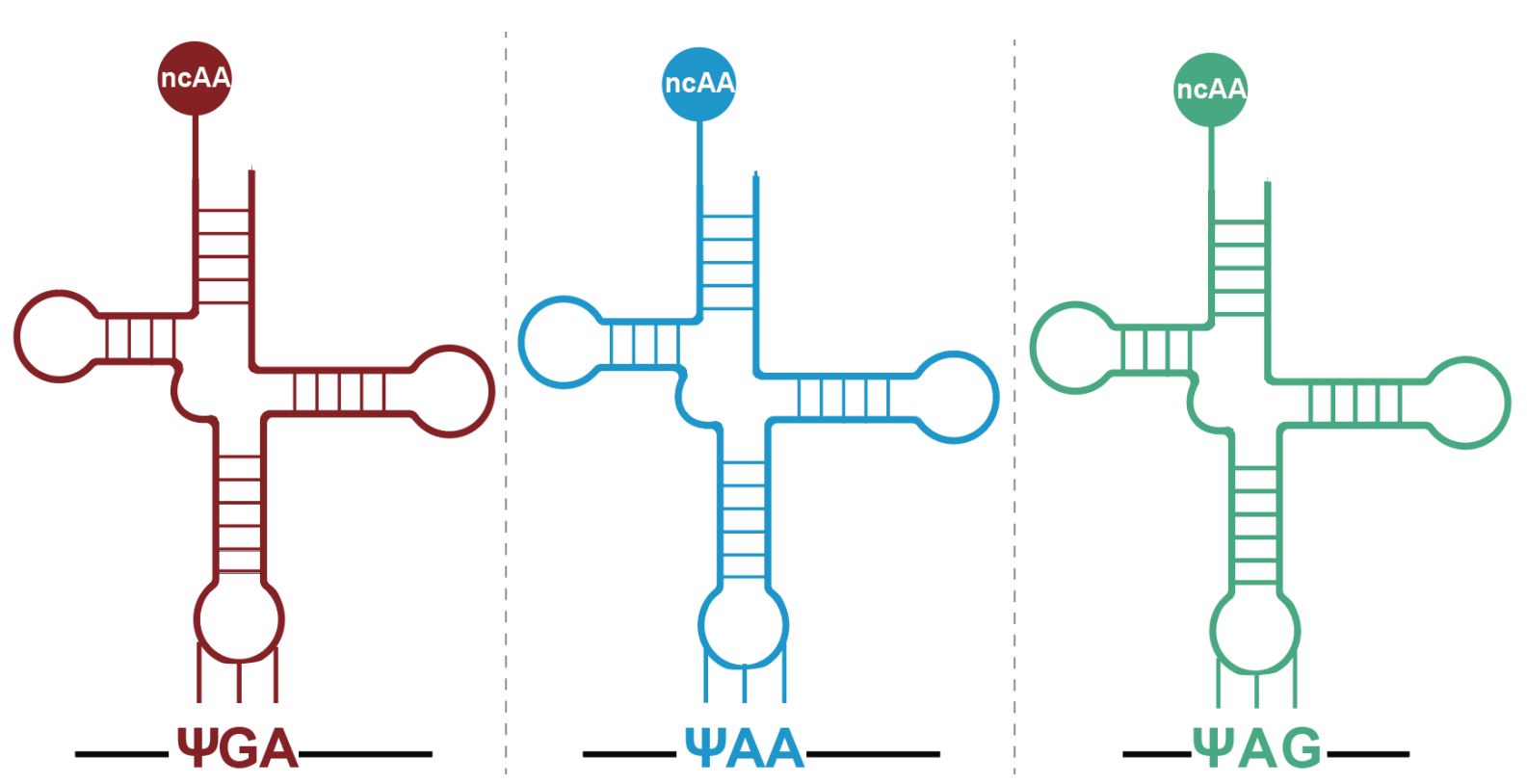
图3. 三对 ΨCodon: (ΨCodon)-tRNAPyl 具有专一性,两两正交。
RCE系统的应用
研究团队通过 RCE 系统实现了蛋白质功能的精准调控。以 Src 激酶为研究对象,研究人员在其催化活性中心特异性插入含反式环辛烯基团的非天然赖氨酸(TCOK),成功构建了化学可激活的激酶突变体。实验证实,该系统能高效识别 ΨGA 密码子并准确插入 TCOK ,形成活性被暂时"锁定"的激酶。当加入四嗪(Tz)分子后,TCOK 被剪切恢复为天然赖氨酸,从而释放激酶活性。通过免疫印迹和磷酸化蛋白质组学分析,研究团队验证了该系统的精确调控能力,证明 RCE 系统特异、高效地实现功能性非天然氨基酸的插入。
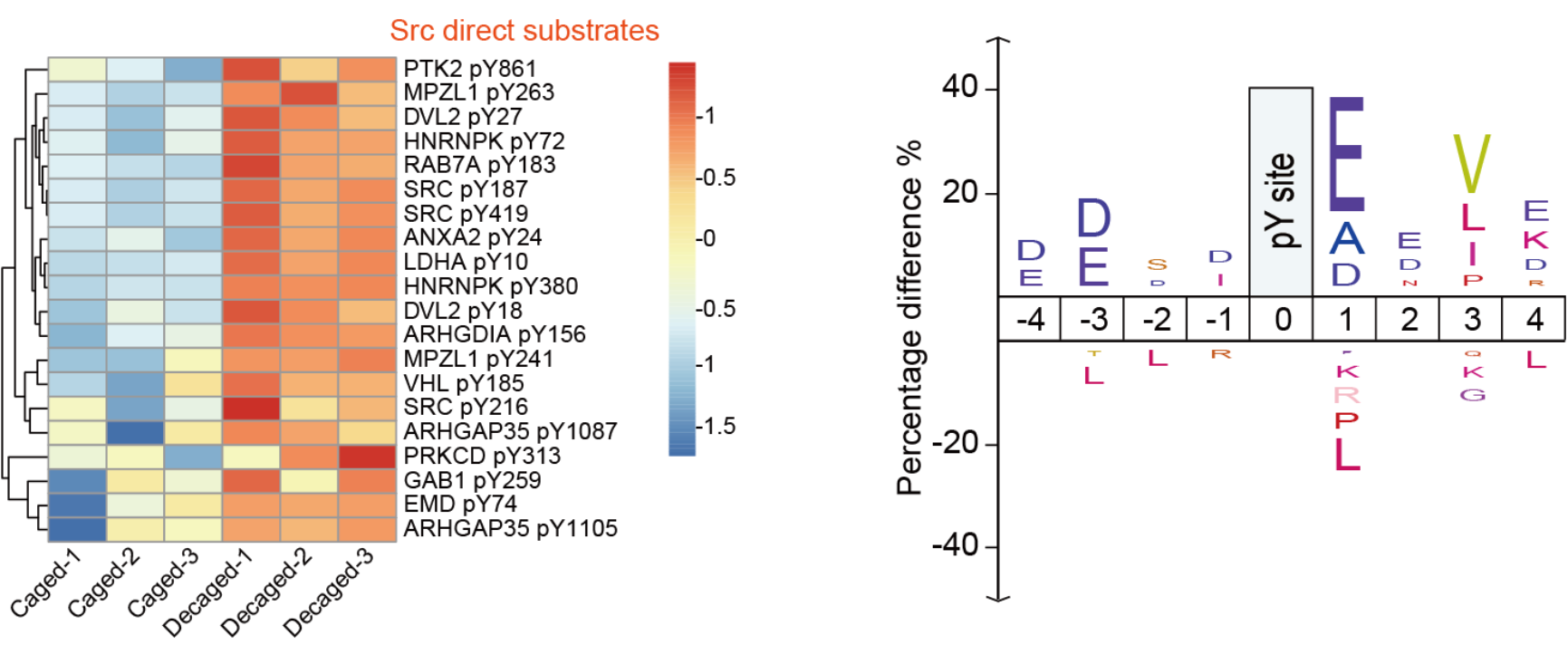
图4. RCE系统特异、高效地实现功能性非天然氨基酸的插入从而调控Src激酶活性。
研究团队进一步验证了 RCE 系统的普适性,成功利用 ΨAG 密码子及其对应 tRNA 解码器,在肿瘤抑制蛋白 p53 的关键核定位序列中精准插入TCOK非天然氨基酸。实验证实,该系统能实现对 p53 蛋白入核过程的时空特异性化学操控。这一突破性进展不仅体现了RCE系统在蛋白质工程中的应用前景,更为合成生物学和生物医学研究提供了全新的工具平台。
本研究通过开发 ΨGA、ΨAA 和 ΨAG 三种新型RNA 密码子,建立了具有高度特异性的遗传密码扩展系统。该系统可以实现非天然氨基酸的精准插入,并支持具有化学成键/断键活性的功能基团整合,为蛋白质在活细胞内的定制合成和工程改造提供了全新策略。这项研究不仅突破了天然遗传密码的限制,更为合成生物学和精准医学研究提供了革命性的工具平台,为未来设计人工生物系统和开发新型生物疗法奠定了重要基础。
陈鹏教授、伊成器教授为本文的共同通讯作者。北京大学前沿交叉学科研究院2019级博士研究生刘江乐、北京大学生命科学学院博士后闫学青博士、前沿交叉学科研究院2020级博士研究生乌浩为本文的共同第一作者。
本工作获得科技部、农业部、国家自然科学基金委、北京市科学技术委员会、北京分子科学国家研究中心、新基石基金会和科学探索奖等项目的支持。
此外,北京大学核糖核酸北京研究中心和北京科学智能研究院温翰研究员提供了核酸分子理论计算的专业指导意见;上海科技大学刘如娟课题组也在核酸分子生化检测方面做出了重要贡献。
原文链接:https://doi.org/10.1038/s41586-025-09165-x
排版:高杨
审核:肖熠,刘志博