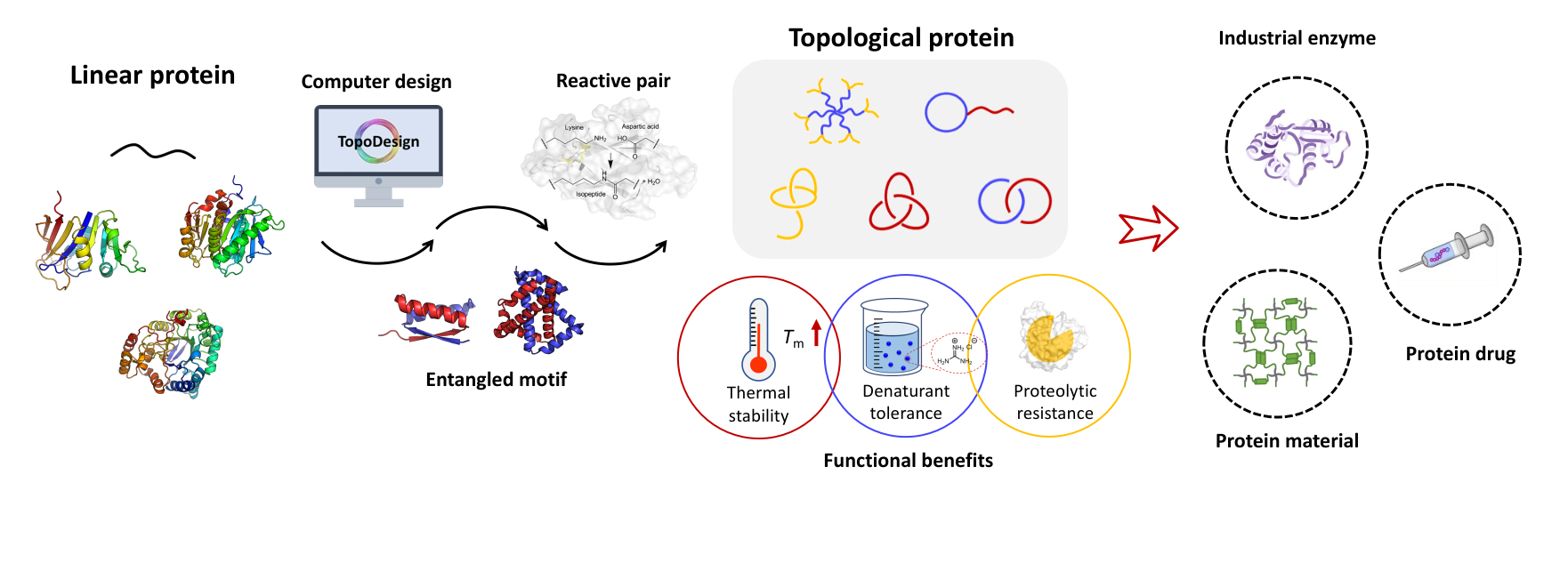
Recent Highlights
Contact us
张文彬课题组
地址:北京市海淀区成府路202号
北京大学化学与分子工程学院
邮编:100871
电话:010-62766876
电邮:wenbin@pku.edu.cn

请扫以上二维码关注我们课题组的公众号。
我们将定期推送组会每周精读和泛读文献介绍以及课题组的最近新闻!
--------------------------------------------
News
Systematic Discovery and Feature Analysis of Intertwined Symmetric Protein Motifs for Topology Engineering
Lianjie Xu, Puqing Deng, Hanyu Gao, Wen-Bin Zhang
Giant 2024, xx, xxxx.
https://doi.org/10.1016/j.giant.2023.100226
内容简介
近年来,利用天然存在或人工设计的蛋白质缠结基元,通过“组装-反应”协同策略,研究者合成了一系列人工拓扑蛋白质,包括纽结、套索和链环等不同类别,拓扑工程成为调控蛋白质结构与功能的一个重要手段。然而目前研究者所使用的缠结基元仍十分有限,这也导致目前所能设计与合成的蛋白质拓扑结构仍较为简单。因此,为了拓展可用的蛋白质缠结基元,北京大学张文彬教授课题组对PDB中的对称蛋白质组装体(包括Cn、Dn、tetrahedral、octahedral和icosahedral等不同对称性类别)进行了系统检索,根据高斯连接数(GLN)、包埋表面积(BSA)、N-C端距离(dN-C)等参数自动筛选并辅以人工校验,建立了对称蛋白质缠结基元数据库,并进一步结合分子功能的分析,揭示了蛋白质链缠结可能的生物学意义。蛋白质缠结基元数据库的建立大大拓展了复杂拓扑的设计可能性,增进了对蛋白质链缠结行为的理解,为人工拓扑蛋白质材料在酶工程和生物医药等方面的应用奠定了基础。
研究背景
受限于生物合成的翻译机制,蛋白质链天然具有线型主链结构。然而,近年来研究者陆续发现具有非线型主链的天然蛋白质,包括环状、纽结、套索和链环等不同的拓扑类别。对于这些天然拓扑蛋白质的研究表明,非线型拓扑常表现出稳定性优势,这也启发研究者设计与合成人工拓扑蛋白质,并基于拓扑蛋白质发展新颖的生物医药材料。与化学拓扑或DNA拓扑的设计一样,蛋白质拓扑结构的实现主要依赖对蛋白质链的空间关系和连接关系的精确调控。基于天然或人工设计的缠结基元和可基因编码的化学偶联工具分别介导蛋白质链的空间关系和连接关系,研究者通过“组装-反应”协同策略,已经成功合成了一系列人工拓扑蛋白质。然而目前可供使用的缠结基元非常有限且多为二聚基元,这导致所合成的人工拓扑蛋白质的复杂度仍相对简单。因此,建立缠结基元数据库对于拓展人工拓扑蛋白质的设计可能性,加深对蛋白质链缠结的生物学意义和拓扑蛋白质的构效关系的理解,具有重要的科学意义。
本文亮点
1. 针对人工拓扑蛋白质的设计需求,建立了对称缠结基元数据库,大大拓展了复杂蛋白质拓扑的设计空间。
2. 通过对C2基元的GLN、BSA、dN-C以及分子功能的分析,揭示了蛋白质链缠结在提高稳定性、传导力信号和调控结构域距离与取向等方面可能具有的生物学意义。
图文导读
北京大学张文彬教授课题组长期致力于人工拓扑蛋白质的设计、合成与应用研究(Chin. J. Chem. 2023, 41, 2873)。近年来基于“组装-反应”协同策略,通过天然二聚缠结基元p53dim及其变体和人工重接线CnaB2与RrgA结构域设计的正交主动模板,成功合成了一系列人工拓扑蛋白质,并且证明了非线型拓扑在提高蛋白质稳定性方面具有显著优势(图1)。
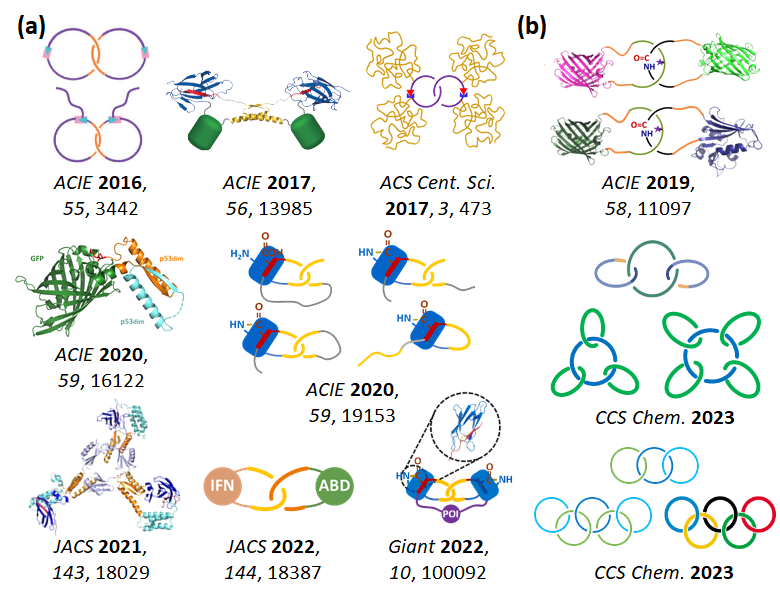
图1 人工拓扑蛋白质。(a) 基于天然缠结基元作为模板合成的各类蛋白质拓扑结构;(b) 基于人工设计的主动模板合成的各类蛋白质拓扑结构。
基于已取得的研究成果,该团队进一步设想合成更为复杂的蛋白质拓扑结构,但这依赖于所使用的缠结基元在对称性、缠结程度和N-C端取向等几何参数上的多样性。为此,张文彬课题组对PDB中的对称蛋白质组装体进行了系统的检索,使用GLN评估链缠结程度,使用BSA评估组装体稳定性,使用dN-C评估链关环的难易程度,通过几何参数的自动筛选辅以人工校验,建立了适配蛋白质拓扑工程的对称缠结基元数据库(图2)。
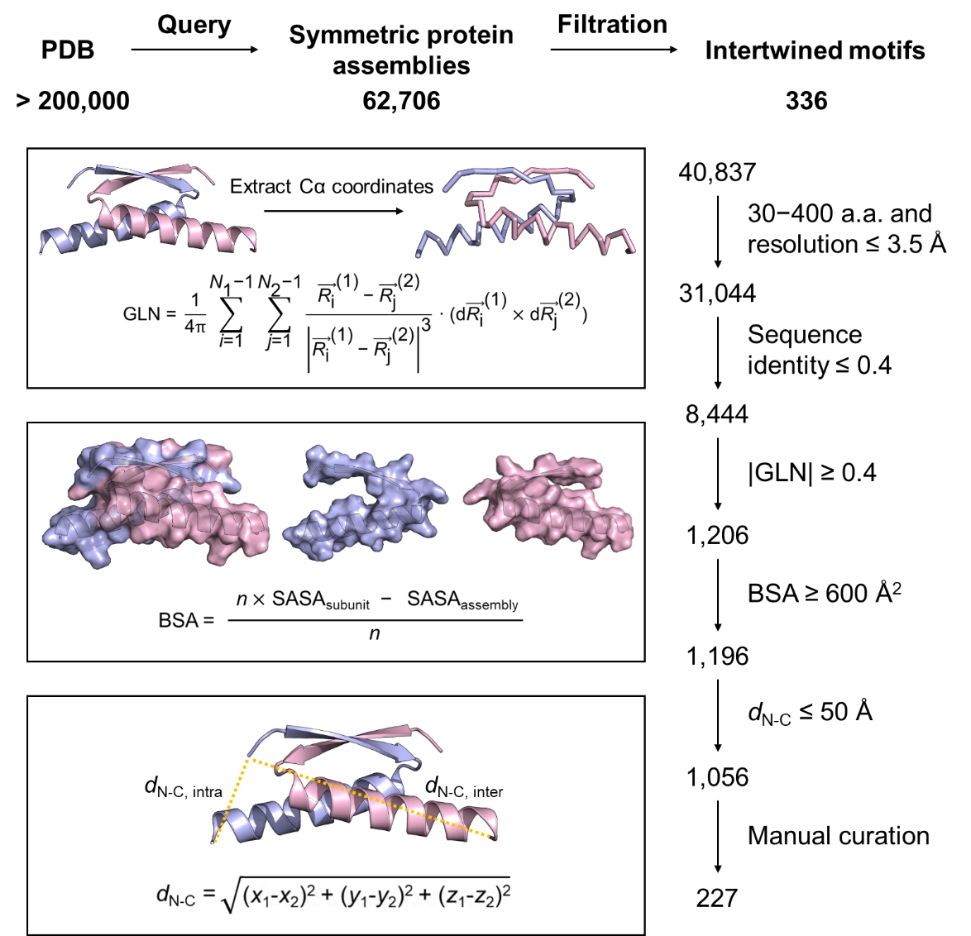
图2. 蛋白质缠结基元的筛选流程(以C2基元为例)。
图3展示了筛选出的具有设计潜力的部分对称蛋白质缠结基元,相较于此前已知仅有的数个缠结基元,缠结基元的多样性和复杂度得到显著提高。
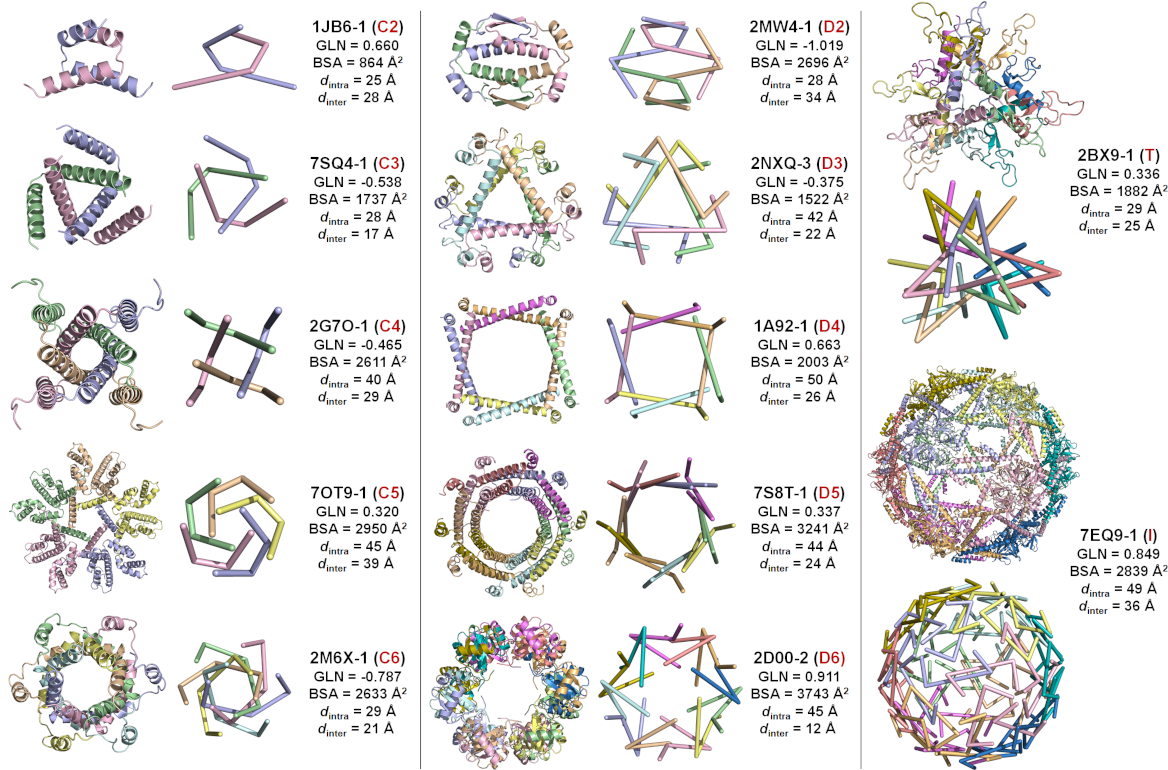
图3. 筛选出的部分具有设计潜力的对称蛋白质缠结基元。
为了增进对蛋白质链缠结的生物学意义的理解,作者对C2组装体的分子功能进行分析,结果表明链缠结程度较高的C2基元的功能集中于特定类别,例如DNA结合蛋白、马达蛋白等,对于这些功能蛋白的深入分析揭示了蛋白质链缠结在提高组装体稳定性、传导力信号和调控结构域距离与取向等方面扮演的重要角色(图4)。
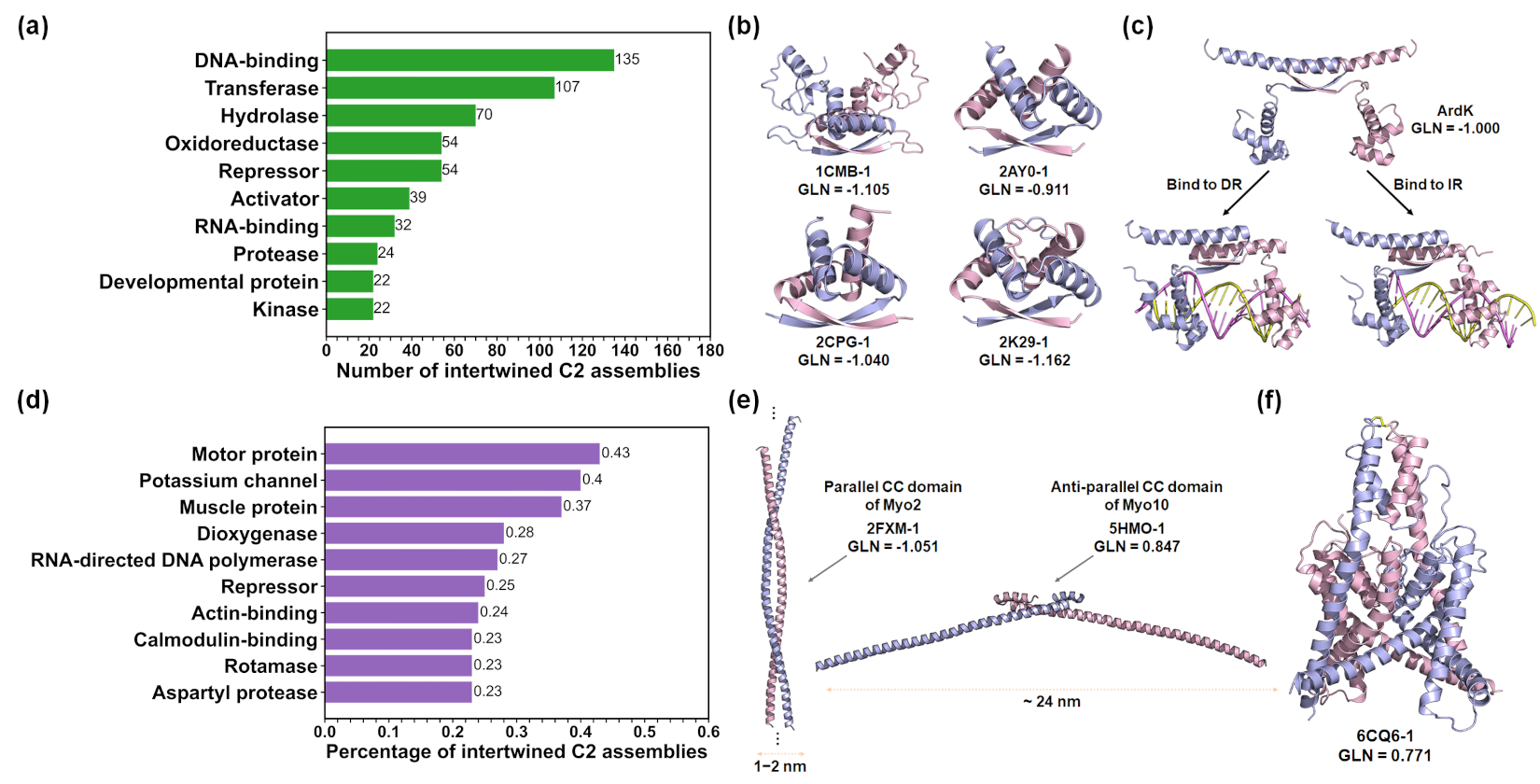
图4. C2缠结基元的功能分析。
利用这些具有不同对称性的缠结基元,可以显著增加所能合成的拓扑结构的种类和复杂度,例如图5中所展示的412和812链环、31和52纽结。可以预见的是,当把不同的缠结基元组合使用时,拓扑结构的复杂度将会得到进一步的提高,甚至比肩大自然的创造。这类复杂蛋白质拓扑组装体在生物医药领域将具有诱人的应用前景。
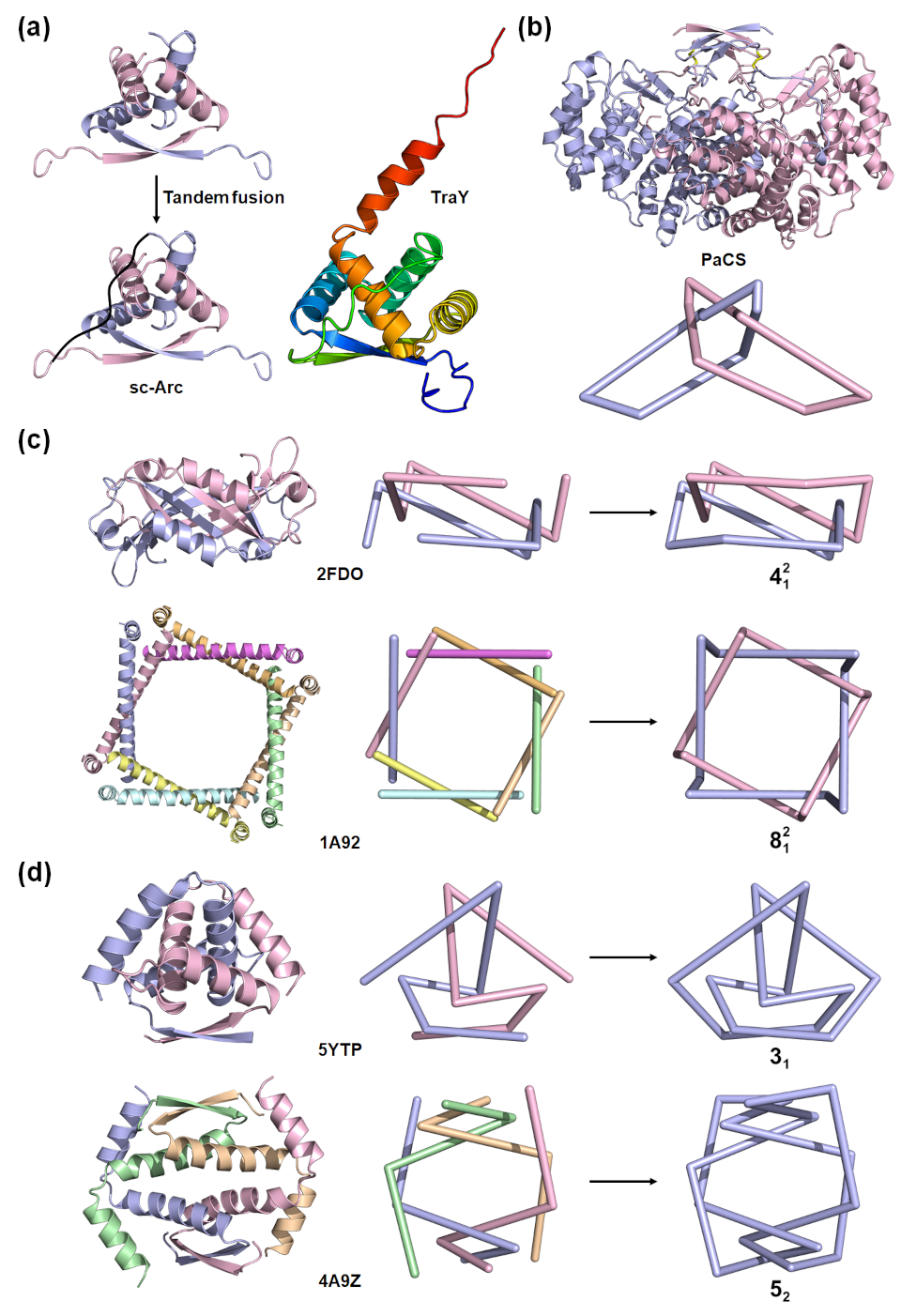
图5. 基于蛋白质缠结基元设计不同的链环和纽结拓扑结构。
总结而言,本研究为复杂蛋白质拓扑结构的人工设计与合成提供了丰富的构筑基元,加深了对于蛋白质链缠结行为的理解,为提高拓扑蛋白质的结构和功能多样性奠定了基础。
作者介绍
许连杰(本文第一作者)
2023年获北京大学高分子化学与物理博士学位,之后在北京大学博雅博士后项目支持下从事博士后研究工作。主要致力于复杂蛋白质拓扑结构的设计、合成与应用研究。
邓埔清(本文第二作者)
香港科技大学化学与生物工程在读博士生,获香港科技大学红鸟博士奖学金。主要致力于将深度学习用于蛋白缠结基元的发掘和设计,以指导拓扑蛋白的合成。
高寒宇(本文通讯作者)
香港科技大学助理教授。主要研究方向是计算机辅助的聚合物合成和材料设计,通过结合反应动力学、分子动力学和机器学习的建模手段,探究合成高分子和天然高分子的构效关系,从而更好的控制材料性能,并促进新材料的开发。
课题组主页:
https://hanyugao.com/
张文彬(本文通讯作者)
北京大学博雅特聘教授,博导,2019年获国家杰出青年科学基金。自独立开展工作以来,以“精密结构高分子”为中心,对高分子的设计、合成和自组装做了积极的尝试和深入的研究,致力于通过结合生物大分子和合成大分子的设计理念和独特基元,发展具有精密结构的非传统高分子,实现对其化学结构和物理结构的精准控制,以发展相应的功能材料。
课题组主页:
https://www.chem.pku.edu.cn/zhangwb/